DNA N6-甲基腺嘌呤(6mA)近年来在真核生物中陆续被鉴定和发现,成为表观遗传学领域的研究热点。在人、小鼠、线虫、拟南芥及单细胞的四膜虫、衣藻和早期分化的真菌中,调控6mA的甲基化酶或去甲基化酶已被系统鉴定和研究[1-7],但有关6mA在真核生物中的功能和调控机理的探索才刚刚起步。
2025年1月,我校海洋生物多样性与进化研究所高珊课题组与南加州大学刘一凡课题组合作在PNAS(《美国科学院院刊》)杂志发表题为“Dual modes of DNA N6-methyladenine maintenance by distinct methyltransferase complexes”(DNA N6-腺嘌呤甲基化酶复合体的双重调控模式)的研究成果。这项研究以单细胞真核生物-四膜虫为模式材料,首次发现了两种不同的6mA甲基化酶复合体,并阐明了它们在维持性甲基化过程中的协同作用。这一双重调控机制确保了DNA复制后6mA甲基化快速、准确的恢复。

真核生物6mA在基因转录调控和染色质结构塑造中发挥着重要作用。团队前期以单细胞真核模式生物四膜虫为研究材料,系统解析了6mA的分布模式[6]、甲基转移酶[7]及其在细胞生长发育中的功能和调控通路[8],并发现6mA以经典的半保留方式进行稳定遗传[9]。然而,特异性分布的6mA位点如何精准且高效地传递给后代,尚不清楚。
该工作鉴定到了两种以6mA甲基化酶AMT1为核心的复合体:AMT6复合体和AMT7复合体(图1A)。两个复合体在多个方面表现出显著差异:在甲基化酶活性方面,AMT7复合体相较于AMT6复合体具有更高的酶活和更强的催化连续性(图1B)。在分布模式方面,AMT7显示出更强的染色质结合力,并呈现基因区倾向性分布;而AMT6的染色质结合能力较弱,且分布模式无显著倾向。在体内调控方面,AMT7敲除株系的6mA水平下降明显,且出现生长缓慢以及巨大伸缩泡等表型,而AMT6敲除株系的上述表型均较为微弱(图C-D)。值得注意的是,两个复合体之间存在一定的竞争性关系:AMT1与复合体的另外两个组分蛋白AMTP1和AMTP2形成一个subcomplex,通过AMT6和AMT7的竞争性组装形成有功能的完整复合体。
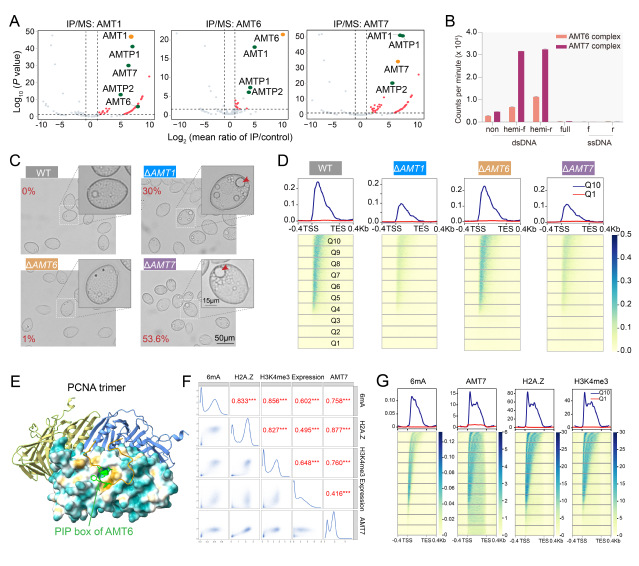
图1. 两种不同的6mA甲基化酶复合体(AMT6复合体和AMT7复合体)。(A)6mA甲基化酶复合体组分蛋白AMT1、AMT6及AMT7的IP-MS结果。(B)两种复合体的酶活检测。(C)AMT1和AMT7敲除后表现出伸缩泡异常增大。(D)AMT1、AMT6、AMT7敲除后的6mA水平及基因组分布情况。(E)AF3预测显示PCNA与AMT6在PIP box处相互作用。(F)6mA、AMT7、H2A.Z、H3K4me3及转录的相关性分析。(G)6mA、AMT7、H2A.Z及H3K4me3在基因区的分布。
两个复合体都倾向于催化半甲基化的DNA底物,符合其作为维持性甲基化酶的特征。AMT6复合体被复制关键蛋白PCNA招募(图1E),主要作用是促进DNA复制后维持甲基化的启动。而AMT7复合体与组蛋白变体H2A.Z和组蛋白翻译后修饰H3K4me3等表观遗传标记密切相关(图1F-G)。它由这些转录相关的表观遗传途径招募,负责绝大多数位点的维持性甲基化。通过分别识别复制和转录通路的信号,两个复合体共同实现6mA位点稳定高效的遗传。
AlphaFold3预测发现(图2),AMT6/AMT7复合体与DNA结合后会发生明显的结构变化,从多变的开放形式转变为单一的封闭形式(图2A-F)。在封闭状态下,复合体呈现由AMT6/AMT7、AMT1和AMTP1共同包裹双链DNA的钳状结构(图2D, F)。两种复合体的蛋白交联实验也证实了这一结果(图2G-H)。上述结果表明,AMT6/AMT7复合体的维持甲基化涉及多个步骤,从两种复合体的竞争性组装开始,到最终形成稳定的封闭结构,从而协同进行6mA甲基化的调控。
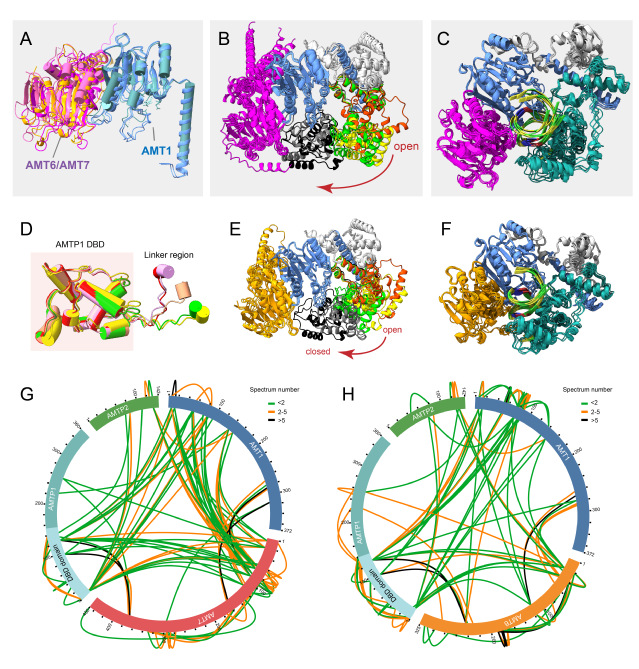
图2. 两种6mA甲基化酶复合体的AF3(AlphaFold3)结构预测。(A)AMT6和AMT7复合体的AF3预测模型。(B)AMT7复合体从开放到关闭的动态转换。(C)AMT7复合体与DNA结合后的稳定状态。(D)AMTP1的DNA结合结构域(DBD)是稳定的,而接头区域表现出高度的灵活性。(E)AMT6复合体的从开放到关闭的动态转换。(F)AMT6复合体与DNA结合后的稳定状态。(G)AMT7复合体的蛋白交联结果。(H)AMT6复合体的蛋白交联结果。
该工作揭示了四膜虫中的6mA维持甲基化能够整合来自复制和转录的信号,通过双模式系统进行多重识别实现精确和快速的甲基化。该工作揭示了6mA位点精准且高效地传递的机制,解析了转录与复制在表观遗传调控中的协同作用,进一步推动了我们对甲基化调控机制的理解(图3)。
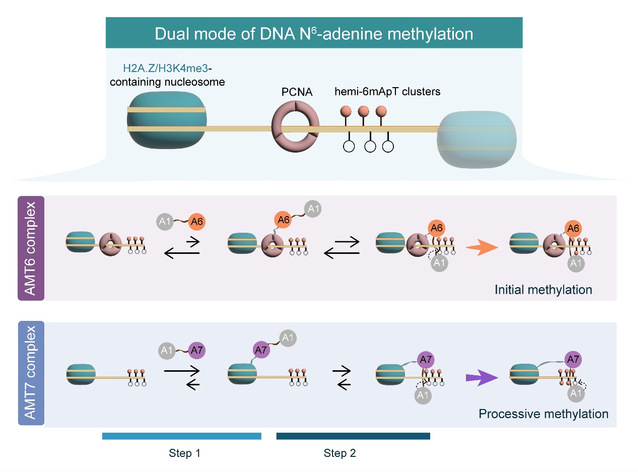
图3. 两种6mA甲基化酶分工协作的模式图。
该研究由进化所高珊课题组与南加州大学刘一凡课题组共同完成。高珊课题组的副教授王媛媛、博士生南北、叶飞、张喆和刘一凡课题组博士后杨文涛为该文章的共同第一作者。高珊教授、刘一凡副教授和王媛媛副教授为文章的通讯作者。云南大学章文信副教授,青岛华大基因研究院王丹彤博士,高珊课题组博士毕业生潘博和段丽丽、博士生魏帆、李海程、牛俊骅、鞠艾利和博士后刘永强对本文亦有重要贡献。
原文链接:
https://www.pnas.org/doi/10.1073/pnas.2413037121
参考文献:
[1]Fu Y, Luo GZ, Chen K, Deng X, Yu M, Han D, Hao Z, Liu J, Lu X, Doré LC. N6-methyldeoxyadenosine marks active transcription start sites in Chlamydomonas. Cell. 2015, 161(4):879-92.
[2]Greer EL, Blanco MA, Gu L, Sendinc E, Liu J, Aristizábal-Corrales D, Hsu CH, Aravind L, He C, Shi Y. DNA methylation on N6-adenine in C. elegans. Cell. 2015, 161(4):868-78.
[3]Zhang G, Huang H, Liu D, Cheng Y, Liu X, Zhang W, Yin R, Zhang D, Zhang P, Liu J. N6-methyladenine DNA modification in Drosophila. Cell. 2015, 161(4):893-906.
[4]Mondo SJ, Dannebaum RO, Kuo RC, Louie KB, Bewick AJ, LaButti K, Haridas S, Kuo A, Salamov A, Ahrendt SR. Widespread adenine N6-methylation of active genes in fungi. Nat Genet. 2017, 49(6):964-8.
[5]Seidl MF. Adenine N6-methylation in diverse fungi. Nat Genet. 2017, 49(6):823-4.
[6]Wang Y, Chen X, Sheng Y, Liu Y, Gao S. N6-adenine DNA methylation is associated with the linker DNA of H2A.Z-containing well-positioned nucleosomes in Pol II-transcribed genes in Tetrahymena. Nucleic Acids Res. 2017, 45:11594-606.
[7]Wang Y, Sheng Y, Liu Y, Zhang W, Cheng T, Duan L, Pan B, Qiao Y, Liu Y, Gao S. A distinct class of eukaryotic MT-A70 methyltransferases maintain symmetric DNA N6-adenine methylation at the ApT dinucleotides as an epigenetic mark associated with transcription. Nucleic Acids Res. 2019, 47(22):11771-89.
[8]Pan B, Ye F, Li T, Wei F, Warren A, Wang Y, Gao S. Potential role of N6-adenine DNA methylation in alternative splicing and endosymbiosis in Paramecium bursaria. iScience. 2023, 26(5):106676.
[9]Sheng Y, Wang Y, Yang W, Wang XQ, Lu J, Pan B, Nan B, Liu Y, Li C, Song J, Dou Y, Gao S, Liu Y. Semi-conservative transmission of DNA N6-adenine methylation in a unicellular eukaryote. Genome Res. 2024, 34:740-56.


