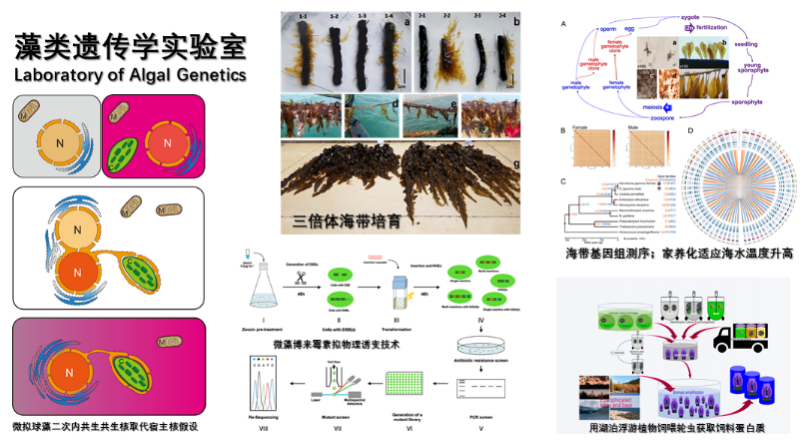
研究方向:
(1)海带遗传资源创制;适应气候变化机制;遗传改良
(2)微藻遗传变异创制;进化生物学分析;遗传改良;微藻与饲料蛋白生产

杨官品 教授 博士生导师
E-mail: yguanpin@ouc.edu.cn
Tel: 132 0641 2039
教育经历
1989-9至1992-12,华中农业大学,作物遗传改良国家重点实验室,博士
1985-9至1988-12,华中农业大学,农学系,硕士
1981-9至1985-7,湖北大学,生物系,学士
科研与学术工作经历
2000-12至现在,中国海洋大学,海洋生命学院,教授
2002-2至2000-11,中国海洋大学,海洋生命学院,副教授
1996-7至2000-2,湖北大学,生命学院,教授(聘)
1995-7至1996-7,华中农业大学,作物遗传改良国家重点实验室,副教授
1993-3至1995-7,弗吉利亚理工学院,作物科学系,洛克菲勒基金会博士后
1992-12至1993-3,华中农业大学,作物遗传改良重点实验室,讲师
1988-9至1992-12,华中农业大学,作物遗传改良国家重点实验室,在职博士
1987-9至1988-9,华中农业大学,作物遗传改良国家重点实验室
研究兴趣
1、藻遗传图谱构建、基因组测序、遗传育种、基因从头克隆
2、微藻-微生物共生、共栖、敌害关系、群落演替、生物质和高附加值积累
正在承担的课题
1、国家重点研发计划(蓝色粮仓)子课题“海洋微拟球藻耐低盐性状的遗传基础”,2018.12-2022.12. 2018YFD0900305. 在研
2、国家重点研发计划(蓝色粮仓)子课题 “海洋微拟球藻耐低盐性状的遗传基础”,2018.12-2022.12. 2018YFD0901506. 在研
3、山东省支持青岛海洋科学与技术试点国家实验室专项海洋科技资金重大科技创新工程项目 子课题“海洋单胞藻基因组学及环境适应机制”2018.12-2010.12. 2018SDKJ0406-3. 在研
近年来承担的课题
1、主持 973前期预研项目“微录球藻(Nannochloropsis sp.) EPA生成途径与酶系表达生态调控研究”, 2005.7-2007.7.
2、主持 863探索项目“海洋微藻合成PUFA关键酶基因研究技术”, 2007.12-2010.12. 2007AA09Z400.
3、主持 国家自然科学基金“海洋浮游植物分子生态学研究”, 2002.1-2004.12. 40176028.
4、主持 国家自然科学基金“鲈鱼基因内微卫星标记连锁图谱构建”, 2005.1-2005.12. 30471318.
5、主持 国家自然科学基金“微拟球藻淡水-海水生长相变分子机制研究”, 2013.1-2017.12.
6、参加 国家自然科学基金“海洋浮游植物群落演替的营养盐响应机制研究”,2010-2013
7、参加 科技攻关计划“鲢鱼新品系选育技术研究”, 2005.7-2006.6. 2004BA526B0108.
8、参加 863计划项目“耐高温海带良种选育技术”, 2004.3-2005.10. 2004AA603210.
9、参加 山东省攻关项目“海带杂种优势预测研究”, 2005-2008. 2005GG3205109.
10、参加 支撑计划课题“鲢新品种选育技术研究”, 2007.10-2010.12. 2006BAD01 A120502.
11、参加 科技支撑计划“海洋微藻工程化高效培养技术集成与示范”,2007-2010。
12、参加 国家支撑计划“能源微藻育种与高效生产关键技术研究与示范”,2012-2015
13、参加 国家863计划“优良特色藻种的筛选和评价体系研究”,2014.1 – 2016.12.
14、参加 国家863计划“基于全基因组信息的鱼类遗传育种”,2012-1015
15、参加 国家863计划“基于全基因组信息的藻类遗传选育”,2012-2015
16、参加 海洋公益性专项“几种重要海洋药用生物种质资源发掘、保藏和利用”,2012-2015
教学情况
2015年-2019,本科生《生物学专业导航课》,学时数4.
2000-2017年,本科生《生物学专业英语》,学时数54.
2000-2017;2020年,研究生学位课《分子生物学》,学时数54.
近期代表论文
1. Guo L; Liang S; Zhang Z; Liu H; Wang S; Pan K; Xu J; Ren X; Pei S; Yang G*; Genome assembly of Nannochloropsis oceanica provides evidence of host nucleus overthrow by the symbiont nucleus during speciation, Communications Biology, 2019, 2: 249.
2. Pan J; Zhang Z; Yu S; Han H; Jiang X; Jiang L; Liang G; Sun J; Li X*; Yang G, Growth, artificial seedling raising and cultivation of Sargassum confusum (Fucales, Phaeophyceae) inhabiting the coast of Shandong Peninsula, China, Journal of Applied Phycology, 2019, 31: 1863–1871
3. Ling S; Guo L; Lin G; Zhang Z; Ding H; Wang Y; Yang G*, Improvement of Nannochloropsis oceanica growth performance through chemical mutation and characterization of fast growth physiology by transcriptome profiling, Chinese Journal of Oceanology and Limnology, 2017, 35(4): 792-802
4. Li X; Zhang Z; Qu S; Liang G; Sun J; Zhao N; Cui C; Cao Z; Li Y; Pan J; Yu S; Wang Q; Li X; Luo S; Song S; Guo L*; Yang G*, Improving seedless kelp (Saccharina japonica) during its domestication by hybridizing gametophytes and seedling-raising from sporophytes, Scientific Reports, 2016, 6: 21255
5. Zhang N; Zhang L; Tao Y; Guo L; Sun J; Li X; Zhao N; Peng J; Li X; Zeng L; Chen J, Yang G*, Construction of a high-density SNP linkage map of kelp (Saccharina japonica) by sequencing Taq I site associated DNA and mapping of a sex determining locus, BMC Genomics, 2015, 16: 189
(1) 国家研发计划(蓝色粮仓)项目(2022YFF1102300)
(2) 山东省重点研发计划项目 (2022LZGC004)
2024 Tian P, Wang W, Li Y, Liang G, Sun J, Liu Y, Yang G, Li X. Independent induction of calluses and nematophytes from exfronds and their potential application for kelp cultivation and germplasm conservation. Journal of Applied Phycology doi: 10.1007/s10811-024-03351-6.
2024 Guo L, Yang G. Pioneering DNA assembling techniques and their applications ineukaryotic microalgae. Biotechnology Advances 70: 108301.
2022 Zhang Z, Guo L, Liu H, Pan X, Liu L, Zong Y, Yang G. Zeocin treatment significantly elevated transformation efficiency of Nannochloropsis oceanica. Journal of Applied Phycology doi: 10.1007/s10811-022-02703-4.
2019 Liang S, Zhang Z, Liu H, Guo L, Sun S, Yang G. Identifying the growth associating genes of Nannochloropsis oceanica by bulked mutant analysis (BMA) and RNA sequencing (BMR-seq). Journal of Applied Phycology 31: 3677-3690.
2019 Guo L, Liang S, Zhang Z, Liu H, Wang S, Pan K, Xu J, Ren X, Pei S, Yang G. Genome assembly of Nannochloropsis oceanica provides evidence of host nucleus overthrow by the symbiont nucleus during speciation. Communication Biology 2: 249.