基因活性与其所处的染色质环境密切相关,解析染色质环境对于理解基因调控的复杂网络至关重要。多细胞真核生物中的DNA N6-甲基腺嘌呤(N6-methyladenine, 6mA)含量极低,利用这一稀缺性,科学家们近年来创新性地利用无序列偏好性的6mA甲基化酶精准引入外源6mA修饰,为破解染色质动态调控密码开辟了新途径。近日,进化所原生动物学团队高珊教授在Cell Press细胞出版社旗下期刊Trends in Genetics发表综述文章“Sequence-independent 6mA methyltransferases for epigenetic profiling and editing”,系统梳理了外源6mA标记技术与三代长读长测序相结合在染色质图谱绘制、蛋白质—DNA互作定位和靶向表观遗传编辑方面的应用,着重分析了其方法学优势,并就当前技术瓶颈和优化前景展开讨论。

无偏好性6mA甲基化酶与三代测序:染色质景观解析的革新
利用表观遗传标记解析染色质环境的研究始于利用5-甲基胞嘧啶(5mC)甲基化酶对染色质进行标记,在体内及体外实验中实现对染色质空间构象的解析,但真核生物中高水平的内源5mC使数据解读变得复杂。6mA作为一种新兴的表观遗传标记,广泛分布于原核生物和低等真核生物,在高等真核生物中含量稀少,这为利用6mA甲基化酶进行染色质标记提供了更低的背景。
Dam是最常用的6mA甲基化酶,多应用于基因组分析及遗传工程技术(如DamID技术),但其GATC的催化序列很大程度上限制了分辨率。相比而言,EcoGII和Hia5是两种无序列偏好性的6mA甲基化酶,催化活性高且底物范围广,已成为6mA表观遗传工程领域的重要工具酶。
随着6mA研究热度的持续攀升,更高精度、更具扩展性的6mA检测技术应运而生。基于6mA抗体的6mA免疫沉淀测序技术(6mA-IP)往往受限于抗体的亲和力和特异性,而基于化学或酶学脱氨转化的方法虽可达到单碱基分辨率,但其转化效率仍有待提升。近年来,以Pacific Biosciences和Oxford Nanopore Technologies为代表的第三代测序技术,分别通过监测DNA聚合酶动力学扰动和离子电流变化,实现了对DNA甲基化修饰的单碱基分辨率直接检测(图1A)。相比二代测序,三代测序无需PCR扩增和DNA片段化处理,可直接检测表观遗传标记并真实地反应单分子异质性,同时克服了二代测序在序列高度重复区域和结构复杂区域的多重比对问题(图1B)。将三代测序技术和外源6mA标记技术相结合,为解析染色质环境、检测蛋白质—DNA互作位点及利用6mA进行表观遗传编辑研究提供了全新视角。
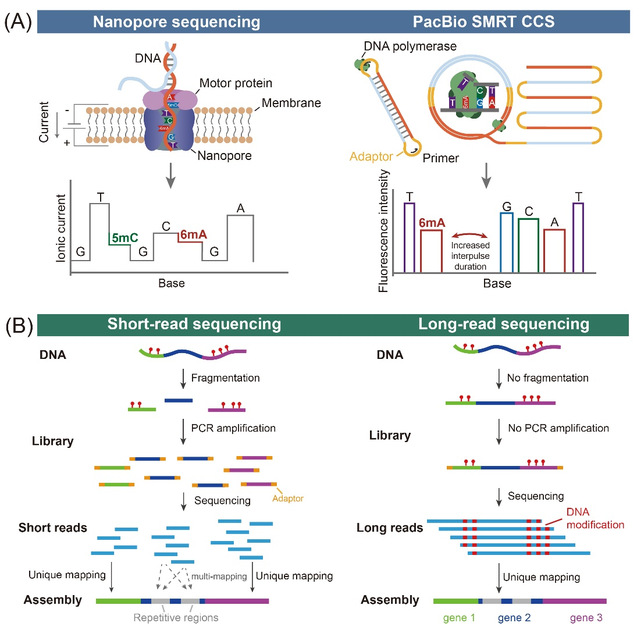
图1 三代测序能够在复杂区域进行DNA甲基化检测
外源6mA标记技术绘制染色质图谱
目前已有多种研究染色质景观的技术方法,如DNase-seq、MNase-seq和ATAC-seq等,但这些依赖于短读长测序的方法都存在固有缺陷。nanoNOMe方法利用GpC 5mC甲基化酶结合纳米孔测序检测了哺乳动物细胞中的核小体占位和染色质开放情况,但基因组中的低GpC密度限制了其空间分辨率。在此基础上,利用EcoGII和Hia5在开放染色质区域无偏好性地标记6mA,并结合三代测序,多个方法被开发出来,实现了单分子、单碱基分辨率下的染色质状态解析,包括核小体占位情况、核小体缺失区域及转录因子结合位点的高精度检测(图2)。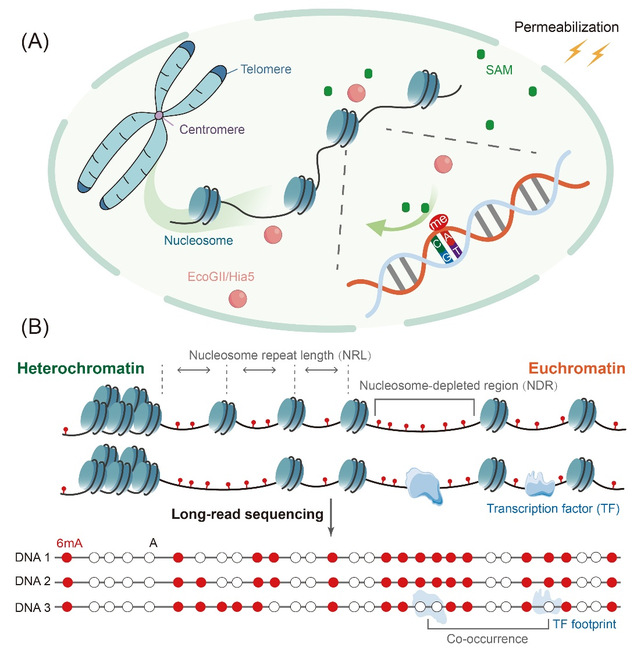
图2 利用6mA甲基化酶和三代测序绘制染色质环境图谱
目前,该技术已在玉米和人类脑组织等多个体系中得到了应用。然而,这类方法目前仍受限于较高的细胞投入量和三代测序较高的成本,酶浓度和反应时间的精准调控对获得最佳结果也至关重要。研究人员正从多方面持续优化该技术,例如,已有报道通过将Tn5转座酶与发夹寡核苷酸结合显著降低了样本细胞的投入量。未来,随着测序成本的下降和酶工程技术的进步,该技术有望在更广泛的生物学研究和临床诊断中发挥重要作用。
外源6mA标记技术揭示蛋白质—DNA互作
解析蛋白质—DNA相互作用有助于揭示染色质调控的时空动态特征。ChIP-seq、CUT&Tag和CUT&RUN等方法应用广泛,但仍存在二代测序的固有问题(图3A)。近年来发展的DiMeLo-seq等技术采用了创新策略:通过抗体孵育通透后的细胞核,利用protein A融合表达的EcoGII或Hia5,在目标蛋白或组蛋白修饰位点附近特异性引入6mA标记;随后借助三代测序技术,可直接在单分子水平获得蛋白结合的位点信息,并可在序列高度重复的着丝粒和亚端粒区域检测到新的蛋白互作位点(图3B)。
除此之外,6mA甲基化酶也可直接与目标蛋白在体内融合表达(图3C)。这种无需抗体的策略有效避免了因抗体表位识别效率不佳而产生的非特异性背景信号,但同时融合表达后目标蛋白的功能与定位也需要进行严格的验证。
同样的,该方法仍存在测序成本较高和细胞起始需求量较大的技术瓶颈。在这一方面,CRISPR介导的特异性片段富集技术为提升检测灵敏度和效率提供了可行思路。另一方面,protein A和抗体之间是非共价结合,在高盐或变性等严苛条件下其稳定性可能受到影响。相比之下,共价标记或基于纳米抗体的策略可实现更高的结合稳定性,为蛋白质靶向染色质标记提供了更优的技术路径。
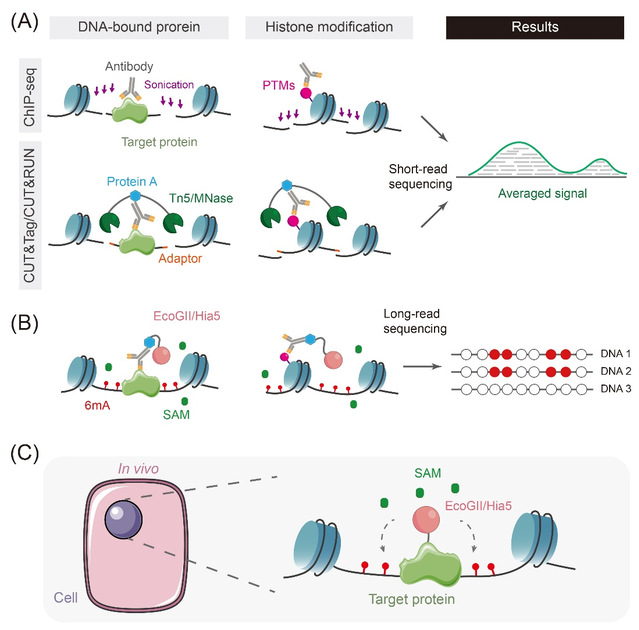
图3 利用6mA甲基化酶和三代测序检测蛋白质—DNA相互作用
6mA甲基化酶介导表观基因编辑
表观基因编辑工程通过在特定基因组区域精确引入或去除表观遗传修饰,实现对基因表达的可编程调控。基于dCas9(丧失核酸酶活性但保留DNA结合能力)系统,可将5mC甲基化酶或去甲基化酶精准导向目标基因,从而实现特定位点的DNA甲基化添加或去除,达到对基因转录状态的精确调控。近年来,6mA甲基化酶在特定位点的表观遗传编辑中崭露头角,已逐步实现局部范围内的高精度基因调控。已有报道称,在家蚕细胞中,6mA甲基化酶METTL4与dCas9融合表达实现了位点特异性甲基化,使靶基因转录显著下调(图4A)。在人类细胞中,将Dam催化结构域与锌指蛋白或dCas9融合,可以特异性甲基化GATC基序;同时将6mA识别蛋白与转录效应因子(如VP64或KRAB)相连,能够实现强效精确的转录调控,并产生可稳定遗传的表观遗传记忆(图4B)。
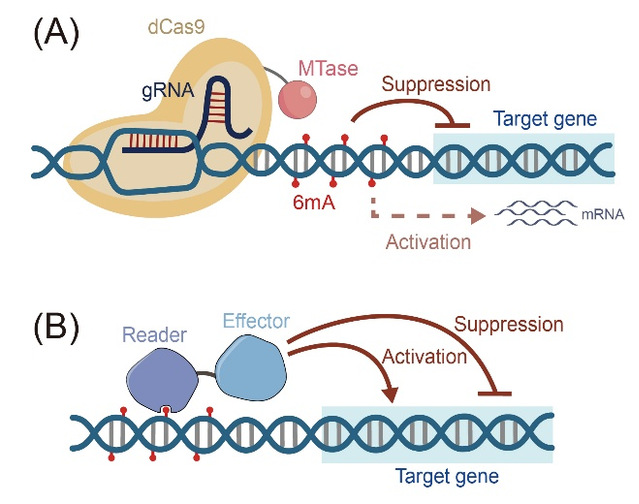
图4 6mA甲基化酶介导表观基因编辑
相比METTL4和Dam, EcoGII和Hia5因具有更高的酶活和更低的序列特异性而展现出广阔的应用前景,但其较高的本底甲基化水平制约了实际应用,亟需进行工程化改造加以优化。可以通过低表达载体、定向突变DNA结合结构域以及改造gRNA等策略提升EcoGII和Hia5的酶活特异性和靶向分辨率,推动6mA介导的真核生物基因调控功能研究。
总结
目前,越来越多的研究利用6mA甲基化酶解析染色质状态和蛋白质—DNA相互作用,并深入探索6mA的生物学功能。但当前该领域仍面临多重挑战,仍需提升靶向系统的精准度,改造具有更高灵敏度与更低背景活性的甲基转移酶,建立可捕获瞬时染色质事件的诱导型标记策略。技术的革新将推动6mA甲基化酶成为更精准的染色质研究工具,并为理解6mA在基因组调控中的功能机制提供新见解。
该文章由进化所原生动物学团队高珊教授课题组撰写。高珊教授课题组博士生张佳晨和张雨苗为该文章的共同第一作者,高珊教授为文章的通讯作者。高珊教授课题组博士生刁静涵和南加州大学刘一凡副教授对本文亦有重要贡献。
原文链接https://www.sciencedirect.com/science/article/pii/S0168952525002367
本文参考文献
1)Stergachis, A. B., Debo, B. M., Haugen, E., et al. 2020. Single-molecule regulatory architectures captured by chromatin fiber sequencing. Science. 368: 1449-1454.
2)Shipony, Z., Marinov, G. K., Swaffer, M. P., et al. 2020. Long-range single-molecule mapping of chromatin accessibility in eukaryotes. Nature Methods. 17(3): 319-327.
3)Abdulhay, N. J., McNally, C. P., Hsieh, L. J., et al. 2020. Massively multiplex single-molecule oligonucleosome footprinting. eLife. 9: e59404.
4)Altemose, N., Maslan, A., Smith, O. K., et al. 2022. DiMeLo-seq: a long-read, single-molecule method for mapping protein-DNA interactions genome wide. Nature Methods. 19(6): 711-723.
5)Weng, Z., Ruan, F., Chen, W., et al. 2023. BIND&MODIFY: a long-range method for single-molecule mapping of chromatin modifications in eukaryotes. Genome Biology. 24(1): 61.
6)Hook, P. W. and Timp, W. 2023. Beyond assembly: the increasing flexibility of single-molecule sequencing technology. Nature Reviews Genetics. 24(9): 627-641.
7)Chen, F., Guo, H., Lan, W., et al. 2023. Targeted DNA N6-methyladenine editing by dCas9 fused to METTL4 in the lepidopteran model insect Bombyx mori. Insect Science. 31(2): 646-650.
8)Nanda, A. S., Wu, K., Irkliyenko, I., et al. 2024. Direct transposition of native DNA for sensitive multimodal single-molecule sequencing. Nature Genetics. 56: 1300-1309.
9)Liu, T. and Conesa, A. 2025. Profiling the epigenome using long-read sequencing. Nature Genetics.
10)Park, M., et al. 2019. Engineering epigenetic regulation using synthetic read-write modules. Cell 176, 227-238.


